研究室概要
ポリアミン調節タンパク質「アンチザイム」に関する研究
はじめに
アンチザイム(AZ)は細胞増殖に必須の「ポリアミン」の細胞内濃度を調節している重要なタンパク質である。ポリアミンは、正電荷を帯びた生理活性物質である。哺乳類の生体内のポリアミンには、プトレシン(Putrescine)、スぺルミジン(Spermidine)。スペルミン(Spermine)の3種が存在している。ポリアミンは、細胞増殖に必須であり、核酸の安定性やmRNAの翻訳促進に寄与している。また、スぺルミジンは、オートファジーの活性化、長寿、記憶などに関与していることが明らかとなってきた。このようにポリアミンは、細胞に必須であるが、その細胞内濃度は、厳密に調節されていなければならない。細胞内のポリアミン濃度は、高すぎても細胞毒性を示し、低すぎてもアポトーシスを誘導し細胞死となる。細胞内には、ポリアミンの濃度を調節する「アンチザイム」と呼ばれるタンパク質が備わっている。
アンチザイムとは
アンチザイム(AZ)は、ポリアミン合成の律速酵素であるオルニチン脱炭酸酵素(ODC)の阻害因子として初めて発見された。AZの発現は、細胞内のポリアミン濃度の上昇に反応して、翻訳フレームシフトと呼ばれるとてもユニークなメカニズムで誘導される。発現したAZはODCに結合し、26SプロテアソームによるODCの分解を促進する。AZはさらに細胞外からのポリアミンの取り込みも阻害する。つまりAZは細胞内のポリアミン濃度を負にフィードバック調節しているのである。AZには3つのタンパク質ファミリーが存在している(AZ1, AZ2およびAZ3)。AZ1とAZ2はほとんどの組織に存在しているが、AZ3は精巣特異的に発現している(表1)。アンチザイムはさらに2つのアンチザイムインヒビター(AZIN)と呼ばれる、ODCにホモロジーがあるがODC活性を持たないタンパク質によって調節されている。表1 アンチザイムファミリー
| AZ1 | AZ2 | AZ3 | |
| 種特異性 | 酵母〜 哺乳動物 |
脊椎動物 | 哺乳動物 |
| 進化的保存 | 低 | 高 | 低 |
| 組織局在 | 全身 | 全身 | 精巣 |
| 発現レベル | 高 | 低 | 低 |
| 翻訳フレームシフト | + | + | + |
| ポリアミンによる発現誘導 | + | + | + |
| ODCへの結合と活性阻害 | + | + | + |
| ODC の分解促進 | |||
| In vitro | + | − | − |
| In vivo | + | + | − |
| AZINへの結合 | + | + | + |
| ポリアミンの細胞内取込阻害 | + | + | + |
1. アンチザイム1
アンチザイム1(AZ1)はアンチザイムファミリーの中では最もよく研究されている。1976年にHellerらによって、動物細胞でポリアミン誘導性のODC阻害因子として報告されている1。AZ1は酵母から哺乳動物まで幅広く存在している(表1)2。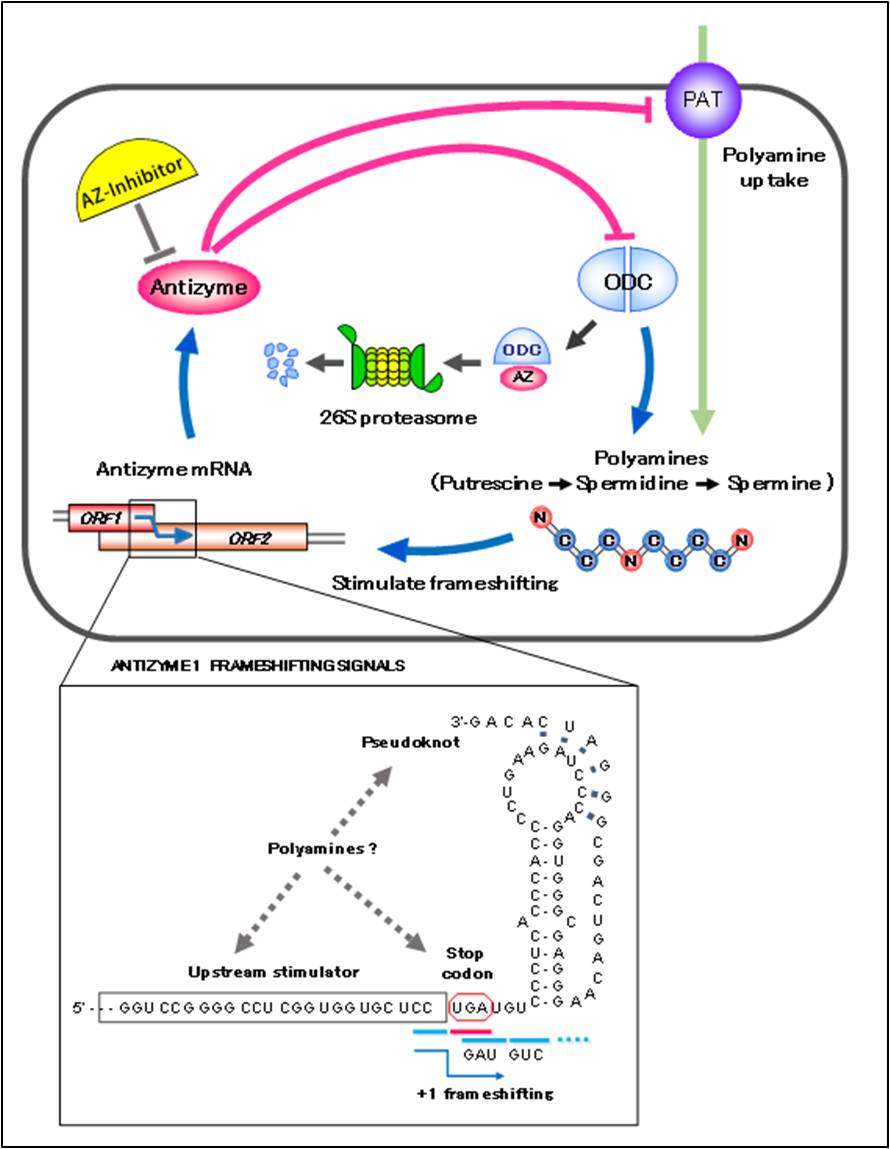
図1 アンチザイムとアンチザイムインヒビターによる細胞内ポリアミンのフィードバック制御
AZ1は単量体ODCに結合しODC活性を阻害するとともにそれを26Sプロテアソームに提示し、ユビキチン非依存的分解を促進する3-6。AZ1自身の分解はユビキチン依存的にプロテアソームで行われることが分かっている7。ODCはAZ1が存在しなくとも26Sプロテアソームで分解されるが(半減期1時間程度)、AZ1が存在した場合 ODCの半減期は数分から十数分となり分解が促進される3。AZ1によるODCの分解促進において2つの構造上重要な領域が存在する。一つはAZ結合領域でアミノ酸の117〜140番目の領域であり8, 9、もう一つはC末端の短い領域で特に末端の5アミノ酸が重要である。C末端領域は、構造的にフレキシブルでAZが結合すると構造が変化しこの構造変化によりODCが26Sプロテアソームにより認識されると考えられている6, 10, 11。AZ1は細胞外からのポリアミンの取り込みを阻害するが12, 13、実際にAZ1が相互作用する輸送体などは見つかっていない。
AZの発現は、ポリアミン誘導性翻訳フレームシフトと呼ばれるとてもユニークなメカニズムで行われる。AZのmRNA上にはオープンリーディングフレームが2つ存在する(ORF1、ORF2)、細胞内のポリアミン濃度が上昇するとAZのmRNA上で翻訳フレームシフトが起こりリボソームの読み枠が1塩基+方向へシフトする。本来認識するはずだったORF1の終止コドンではなくORF2の読み枠でアミノ酸をコードし活性型のAZが合成される2, 20。合成されたAZはODCの単量体に結合し、この複合体が26Sプロテアソームに認識されODCの分解が促進される(図1)。このときAZ自身は分解されず再利用される。このプロテアソームによるODCの分解は、細胞内の多くのタンパク質の分解でおこるポリユビキチン化を必要とせずAZの結合のみで促進される。AZはさらに細胞外からのポリアミンの取り込みも阻害し細胞内のポリアミン濃度を減少させる。つまりAZは細胞内のポリアミン濃度を負にフィードバック調節しているのである。
これまでの研究からAZのmRNA上には+1フレームシフトに必要な3つのシス領域が存在していることがわかっている。フレームシフトサイトから5’上流の50nt、ORF1のストップコドンのUGAおよびフレームシフトサイトから60ntまでの配列で形成されるシュードノット構造の3つである(図1)。これらのシス配列は、フレームシフト効率を各々2.5〜5倍、15〜20倍、2.5〜5倍に上昇させる20-22。酵母では、翻訳途中のアンチザイムのポリペプチド上にリボソームの翻訳とポリペプチドの遊離を抑制させ、リボソームのストールを増加させる部位が存在し、その部位にポリアミンが結合するとリボソームのストールが解除し+1フレームシフト産物の合成を促進しているというモデルが提唱されている23。しかし動物細胞では、ポリアミンがどのように+1フレームシフトを促進しているか、詳細はよくわかっていない。我々は、AZ1〜AZ3の+1フレームシフトメカニズムの解析を一部進行中である。
アンチザイムと相互作用するタンパク質は、AZ1が分解促進するタンパク質はODCのみとされてきたが、最近AZ1によってユビキチン非依存的に分解されるタンパク質がいくつか報告されている(表2)。細胞周期制御因子であるサイクリンD114, 細胞分裂に関連するキナーゼのAuroraA15やMps116、p53のホモログでアミノ末端を欠いているDelta-N(DN)p7317などである。これらのタンパク質に関しては、その分解の詳細や意義に関しての報告はまだ少なく今後の調査が必要であろう。最近我々もAZ1と相互作用する新たなタンパク質としてATPクエン酸リアーゼ(ACLY)を同定した18。この酵素は細胞質のクエン酸からアセチルCoAを合成する酵素である。興味深いことに、AZ1はACLYの分解は全く促進せず逆に活性化しコレステロールや脂肪酸の合成を増加させることが分かった。このことはポリアミン代謝と脂質代謝がリンクしている可能性を示唆するものであり今後の解明が待たれる。
表2 AZ1がユビキチン非依存的に分解を促進するタンパク質(ODC以外)
| Protein names | Protein function | Biological process | Characteristics of degradation |
| ODC | Synthesis of putrescine from ornithine | Polyamine metabolism | AZ1 or AZ2 binds to ODC monomer that is targeted to the 26S proteasome [3, 26] |
| Smad1 | Binding to smad4 Regulation of transcription | BMP signaling pathway | Formation of HsN3-Smad1-AZ1 ternary complex [20]. |
| CyclinD1 | Binding to CDK | Cell cycle | AZ1 binds to Cyclin D1 and accelerates its degradation In vitro and in vivo [14]. |
| Aurora A | Protein Kinase | Mitotic events | Formation of AUKAIP-1-AZ1-Aurora-A ternary complex, and that is target to the proteasome in vivo [15]. |
| Msp1 | Protein Kinase | Cell divition | AZ1 affects the level of Msp1 at the centrosome [16]. |
| DNp73 | Inhibition of both p73 and p53 | Apoptosis | c-Jun dependent DNp73 degradation mediated by AZ1 in vivo [17]. |
| c-Myc | Transcription factor | Cell growth Differentiation survival Apoptosis | AZ2 binds to c-Myc and accelerates its degradation in vivo [19]. |
2. アンチザイム2
AZ2も発現量はAZ1より少ないがAZ1と同様に脊椎動物の組織に広く存在している25。AZ2は、てんかん誘発剤によって神経細胞で発現が上昇する遺伝子の一つとして発見された26 。AZ2はAZ1と同様に細胞内で単量体のODCに結合しその活性を抑制するとともに26Sプロテアソームへ提示しODCの分解を促進させる機能を持っている27。また細胞外からのポリアミンの取り込みも抑制する27, 28。しかしODCとの親和性や分解活性はAZ1の方が高い。不思議なことにAZ2はインビトロの系ではODCの分解促進活性を持たない27。AZ2の細胞内局在はAZ1とは少し異なり主に核である。さらにC末端がリン酸化されることから28、核において独自の機能を有していると考えられる。臨床においては、AZ2のmRNAの発現が高い神経芽細胞腫の患者は生存率が高くなるという正の相関があることが報告されている28。このことはAZ2が神経細胞特異的機能を持っていることを意味しているのかもしれない。我々はごく最近、「アンチザイム2(AZ2)」がん原遺伝子産物のc-Mycと相互作用し、核小体におけるc-Mycのユビキチン非依存的分解を促進することを発見した(図2)19。このことは、アンチザイム2ががん細胞増殖制御できる可能性を意味し、創薬への発展が期待される。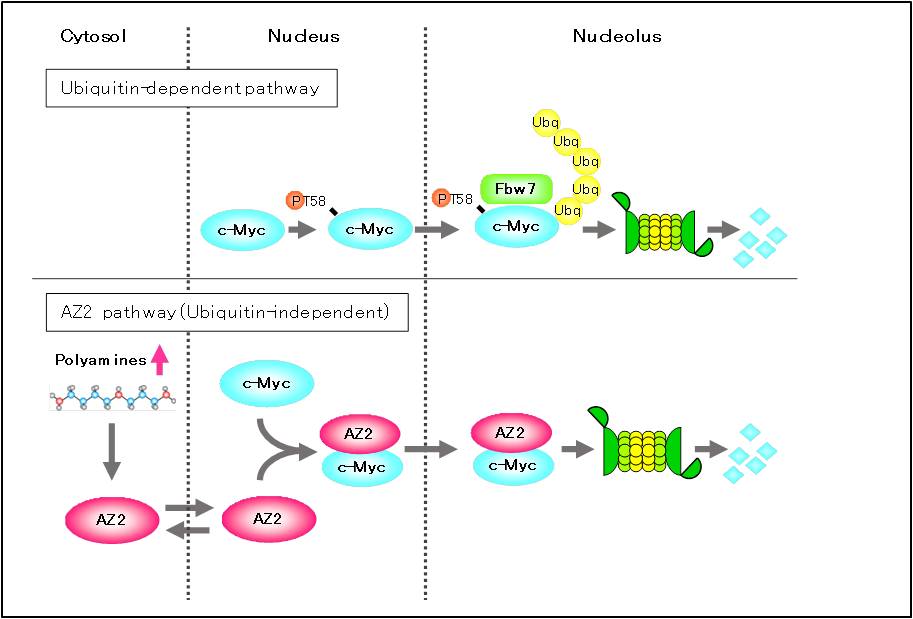
図2 ビキチン依存的分解経路およびAZ2を介したユビキチン非依存的分解経路
3. アンチザイム3
AZ3は雄マウスの生殖細胞特異的タンパク質として同定された26, 30。AZ3のmRNAの発現は精巣に限定され、およそ22 kDaの翻訳産物はAZ1やAZ2と同様に翻訳フレームシフトによって合成されると考えられている。AZ3は細胞内でODCには結合できるが阻害活性は低く30、また分解は促進しない31。AZ3のノックアウトマウスにおいて、メスは正常だがオスはAZ3をノックアウトしても精巣上体や精巣のポリアミン濃度は変わらず、精子も精巣上体から供給されるが、精子の頭部と尾部の連結部位が弱く簡単に分離してしまう31。またAZ3のmRNA翻訳産物の解析から精巣における主な翻訳産物は、翻訳フレームシフト後の22 kDaの産物ではなくORF1のおよそ12 kDaのタンパク質であると報告されている33。今後の解析によりAZ3の機能の詳細が明らかとなるであろう。4. アンチザイムインヒビター1
アンチザイムインヒビター1(AZIN1)はAZに高い親和性で結合しAZの活性を阻害するタンパク質として発見された34。ODCと高い相同性を有しているがODC活性はなく、AZに高い親和性で結合する。AZ-ODC複合体からODCを解離させODCを安定化するとともに、遊離AZを減らしポリアミンの取込みを促進させることにより細胞内のポリアミン濃度を正に調節している35。またAZINにはAZIN1とAZIN2の2つのアイソフォームが存在し、AZIN1はほとんどの組織に普遍的に存在していて細胞増殖とがん化に関与している36 。またKasbekらは、AZIN1はAZ1と共にセントロソームに局在し中心体の複製調節に関与していると報告している16。村上らは、細胞周期に応答してAZIN1の発現や局在が変化することを見出した37。AZIN1とAZ1はセントロソームにおけるMps1(表2参照)の発現を調節している可能性がある。5. アンチザイムインヒビター2
アンチザイムインヒビター2(AZIN2)はODCの活性のないパラログとして2001年に発見された38 。AZIN2は全てのアンチザイム(AZ1〜AZ3)に結合しAZIN1の様にポリアミンの取り込みを促進することができる。AZIN2の局在は脳と精巣に限定され、精巣ではAZIN1の25倍発現しているため39 精巣での機能が予想されていた。しかし最近セロトニン顆粒を含んだマスト細胞にもAZIN2が発現していて、マスト細胞を活性化するとAZIN2の発現が上昇することが報告された40。さらにAZIN2はゴルジ体以降の分泌小胞に局在しその輸送にも関与しているという報告もある41。このようにAZIN2は局在場所に応じて異なる機能をしている可能性がある。参考文献
- Heller, J.S., Fong, W.F. & Canellakis, E.S. Induction of a protein inhibitor to ornithine decarboxylase by the end products of its reaction. Proc Natl Acad Sci U S A 73, 1858-1862 (1976).
- Ivanov, I.P., Gesteland, R.F. & Atkins, J.F. Antizyme expression: a subversion of triplet decoding, which is remarkably conserved by evolution, is a sensor for an autoregulatory circuit. Nucleic Acids Res 28, 3185-3196 (2000).
- Murakami, Y. et al. Ornithine decarboxylase is degraded by the 26S proteasome without ubiquitination. Nature 360, 597-599 (1992).
- Li, X. & Coffino, P. Distinct domains of antizyme required for binding and proteolysis of ornithine decarboxylase. Molecular and cellular biology 14, 87-92 (1994).
- Hayashi, S., Murakami, Y. & Matsufuji, S. Ornithine decarboxylase antizyme: a novel type of regulatory protein. Trends in biochemical sciences 21, 27-30 (1996).
- Coffino, P. Regulation of cellular polyamines by antizyme. Nature reviews. Molecular cell biology 2, 188-194 (2001).
- Gandre, S., Bercovich, Z. & Kahana, C. Ornithine decarboxylase-antizyme is rapidly degraded through a mechanism that requires functional ubiquitin-dependent proteolytic activity. European journal of biochemistry / FEBS 269, 1316-1322 (2002).
- Li, X. & Coffino, P. Regulated degradation of ornithine decarboxylase requires interaction with the polyamine-inducible protein antizyme. Molecular and cellular biology 12, 3556-3562 (1992).
- Hoyt, M.A., Zhang, M. & Coffino, P. Ubiquitin-independent mechanisms of mouse ornithine decarboxylase degradation are conserved between mammalian and fungal cells. J Biol Chem 278, 12135-12143 (2003).
- Murakami, Y., Matsufuji, S., Hayashi, S.I., Tanahashi, N. & Tanaka, K. ATP-Dependent inactivation and sequestration of ornithine decarboxylase by the 26S proteasome are prerequisites for degradation. Molecular and cellular biology 19, 7216-7227 (1999).
- Almrud, J.J. et al. Crystal structure of human ornithine decarboxylase at 2.1 A resolution: structural insights to antizyme binding. J Mol Biol 295, 7-16 (2000).
- Mitchell, J.L., Judd, G.G., Bareyal-Leyser, A. & Ling, S.Y. Feedback repression of polyamine transport is mediated by antizyme in mammalian tissue-culture cells. The Biochemical journal 299 ( Pt 1), 19-22 (1994).
- Suzuki, T. et al. Antizyme protects against abnormal accumulation and toxicity of polyamines in ornithine decarboxylase-overproducing cells. Proc Natl Acad Sci U S A 91, 8930-8934 (1994).
- Newman, R.M. et al. Antizyme targets cyclin D1 for degradation. A novel mechanism for cell growth repression. J Biol Chem 279, 41504-41511 (2004).
- Lim, S.K. & Gopalan, G. Antizyme1 mediates AURKAIP1-dependent degradation of Aurora-A. Oncogene 26, 6593-6603 (2007).
- Kasbek, C., Yang, C.H. & Fisk, H.A. Antizyme restrains centrosome amplification by regulating the accumulation of Mps1 at centrosomes. Mol Biol Cell 21, 3878-3889 (2010).
- Dulloo, I., Gopalan, G., Melino, G. & Sabapathy, K. The antiapoptotic DeltaNp73 is degraded in a c-Jun-dependent manner upon genotoxic stress through the antizyme-mediated pathway. Proc Natl Acad Sci U S A 107, 4902-4907 (2010).
- Tajima, A. et al. Polyamine regulating protein antizyme binds to ATP citrate lyase to accelerate acetyl-CoA production in cancer cells. Biochemical and biophysical research communications 471, 646-651 (2016).
- Murai N, Murakami Y, Tajima A, Matsufuji S. Novel ubiquitin-independent nucleolar c-Myc degradation pathway mediated by antizyme 2. Scientific reports. 2018;8(1):3005-.
- Lin, Y. et al. A novel link between the proteasome pathway and the signal transduction pathway of the bone morphogenetic proteins (BMPs). BMC cell biology 3, 15 (2002).
- Matsufuji, S. et al. Autoregulatory frameshifting in decoding mammalian ornithine decarboxylase antizyme. Cell 80, 51-60 (1995).
- Howard, M.T. et al. Cell culture analysis of the regulatory frameshift event required for the expression of mammalian antizymes. Genes to cells : devoted to molecular & cellular mechanisms 6, 931-941 (2001).
- Petros, L.M., Howard, M.T., Gesteland, R.F. & Atkins, J.F. Polyamine sensing during antizyme mRNA programmed frameshifting. Biochemical and biophysical research communications 338, 1478-1489 (2005).
- Kurian, L., Palanimurugan, R., Godderz, D. & Dohmen, R.J. Polyamine sensing by nascent ornithine decarboxylase antizyme stimulates decoding of its mRNA. Nature 477, 490-494 6 (2011).
- Ivanov, I.P., Gesteland, R.F. & Atkins, J.F. A second mammalian antizyme: conservation of programmed ribosomal frameshifting. Genomics 52, 119-129 (1998).
- Tosaka, Y. et al. Identification and characterization of testis specific ornithine decarboxylase antizyme (OAZ-t) gene: expression in haploid germ cells and polyamine-induced frameshifting. Genes to cells : devoted to molecular & cellular mechanisms 5, 265-276 (2000).
- Chen, H., MacDonald, A. & Coffino, P. Structural elements of antizymes 1 and 2 are required for proteasomal degradation of ornithine decarboxylase. J Biol Chem 277, 45957-45961 (2002).
- Murai, N., Shimizu, A., Murakami, Y. & Matsufuji, S. Subcellular localization and phosphorylation of antizyme 2. Journal of cellular biochemistry 108, 1012-1021 (2009).
- Geerts, D. et al. The polyamine metabolism genes ornithine decarboxylase and antizyme 2 predict aggressive behavior in neuroblastomas with and without MYCN amplification. International journal of cancer. Journal international du cancer 126, 2012-2024 (2010).
- Ivanov, I.P., Rohrwasser, A., Terreros, D.A., Gesteland, R.F. & Atkins, J.F. Discovery of a spermatogenesis stage-specific ornithine decarboxylase antizyme: antizyme 3. Proc Natl Acad Sci U S A 97, 4808-4813 (2000).
- Snapir, Z., Keren-Paz, A., Bercovich, Z. & Kahana, C. Antizyme 3 inhibits polyamine uptake and ornithine decarboxylase (ODC) activity, but does not stimulate ODC degradation. The Biochemical journal 419, 99-103, 101 p following 103 (2009).
- Tokuhiro, K. et al. OAZ-t/OAZ3 is essential for rigid connection of sperm tails to heads in mouse. PLoS genetics 5, e1000712 (2009).
- Ruan, Y., Cheng, M., Ou, Y., Oko, R. & van der Hoorn, F.A. Ornithine decarboxylase antizyme Oaz3 modulates protein phosphatase activity. J Biol Chem 286, 29417-29427 (2011).
- Fujita, K., Murakami, Y. & Hayashi, S. A macromolecular inhibitor of the antizyme to ornithine decarboxylase. The Biochemical journal 204, 647-652 (1982).
- Kitani, T. & Fujisawa, H. Purification and characterization of antizyme inhibitor of ornithine decarboxylase from rat liver. Biochimica et biophysica acta 991, 44-49 (1989).
- Olsen, R.R. & Zetter, B.R. Evidence of a role for antizyme and antizyme inhibitor as regulators of human cancer. Molecular cancer research : MCR 9, 1285-1293 (2011).
- Murakami, Y. et al. The change of antizyme inhibitor expression and its possible role during mammalian cell cycle. Experimental cell research 315, 2301-2311 (2009).
- Pitkanen, L.T., Heiskala, M. & Andersson, L.C. Expression of a novel human ornithine decarboxylase-like protein in the central nervous system and testes. Biochemical and biophysical research communications 287, 1051-1057 (2001).
- Lopez-Contreras, A.J., Ramos-Molina, B., Cremades, A. & Penafiel, R. Antizyme inhibitor 2 (AZIN2/ODCp) stimulates polyamine uptake in mammalian cells. J Biol Chem 283, 20761-20769 (2008).
- Kanerva, K. et al. Expression of antizyme inhibitor 2 in mast cells and role of polyamines as selective regulators of serotonin secretion. PLoS One 4, e6858 (2009).
- Lopez-Contreras, A.J. et al. Subcellular localization of antizyme inhibitor 2 in mammalian cells: Influence of intrinsic sequences and interaction with antizymes. Journal of cellular biochemistry 107, 732-740 (2009).
バナースペース
東京慈恵会医科大学・分子生物学講座
〒105-8461
東京都港区西新橋3-25-8 F棟1階
TEL 03-3433-1111 (内)2276
FAX 03-3436-3897